Naukowcy z IBB współautorami nowej metody selekcji RNA
Nowa metoda przygotowania RNA do sekwencjonowania wysokoprzepustowego
Zespół naukowców pod kierunkiem dr Marii Górnej z Wydziału Chemii UW oraz dr hab. Agaty Starosty z IBB PAN opracował nową metodę przygotowania RNA do sekwencjonowania wysokoprzepustowego. Odkrycie ułatwi badania ekspresji genów dla wielu gatunków grzybów, roślin i mikroorganizmów eukariotycznych.
Metoda opracowana przez zespół dr Marii Górnej polega na wychwyceniu i wzbogaceniu kodujących RNA dzięki wiązaniu (przez białko IFIT1 unieruchomione na złożu) czapeczki na 5’ końcu RNA – tzw. grupy kap 0, która występuje tylko na końcach mRNA.
Analiza przeprowadzona przez zespół dr hab. Agaty Starosty, wzbogaconych w ten sposób RNA pokazała, że nowa metoda może zastąpić komercyjne zestawy stosowane do przygotowywania próbek. Badacze wykazali, że uprzednio znana metoda, oparta na innym białku wiążącym kap, nie nadaje się do próbek RNA drożdży piekarskich – modelowego organizmu w badaniach ekspresji genów i metabolizmu RNA.
Wyniki prac zostały opublikowane w czasopiśmie „Nucleic Acids Research”. W badaniach uczestniczył również dr hab. Rafał Tomecki z Wydziału Biologii UW oraz dr Agnieszka Tudek z IBB PAN. Badania były finansowane m.in. z grantu LIDER NCBiR, First Team FNP oraz EMBO Installation Grant.
Martyna Nowacka+, Przemysław Latoch+, Matylda A Izert, Natalia K Karolak, Rafal Tomecki, Michał Koper, Agnieszka Tudek, Agata L Starosta*, Maria W Górna*, A cap 0-dependent mRNA capture method to analyze
the yeast transcriptome, Nucleic Acids Research, 2022;, gkac903, https://doi.org/10.1093/nar/gkac903
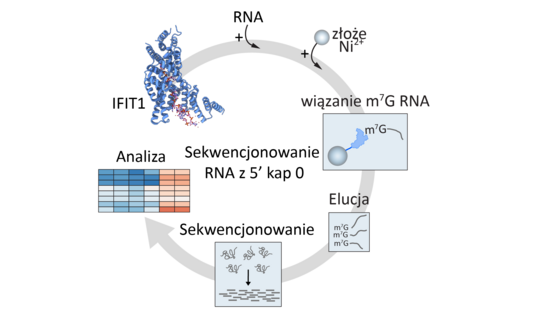
Grafika przedstawia schemat nowej metody wzbogacania mRNA do sekwencjonowania.
Autorzy: dr hab. Agata Starosta i Przemysław Latoch z IBB PAN.


