Badania nad dynamiką procesu deadenylacji mRNA
Naukowcy z IBB PAN, IIMCB, Uniwersytetu w Aarhus oraz Uniwersytetu w Turku przeanalizowali dynamikę deadenylacji ogonów poliadenozynowych (pA) mRNA w drożdżach Saccharomyces cerevisiae w warunkach podstawowych oraz stresowych.
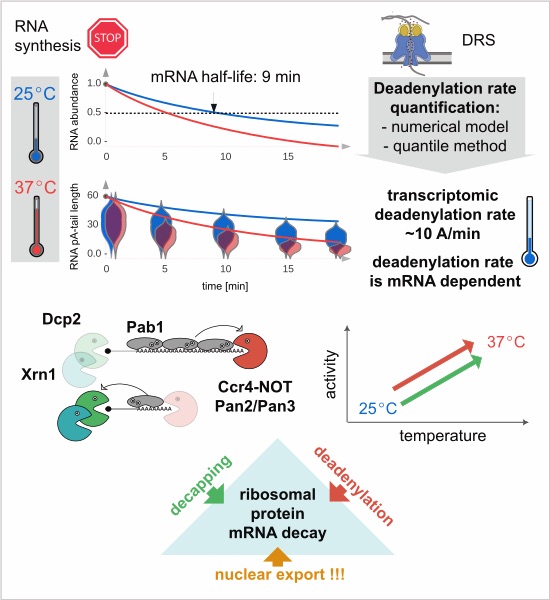
Ogony pA są konserwowaną ewolucyjnie modyfikacją końca 3’ mRNA, która jest wymagana do eksportu transkryptu z jądra do cytoplazmy oraz jego translacji. Przyjęte modele pokazują, że sukcesywne skracanie ogona pA wyznacza czas trwania transkryptu w cytoplazmie. Transkrypty, które uległy znacznej deadenylacji są dekapowane oraz degradowane od końca 5’ przez egzonukleazę Xrn1.
W swej pracy naukowcy z IBB PAN oraz IIMCB stworzyli model deadenylacji transkryptomu oparty o modyfikowaną dystrybucję gamma, dzięki czemu ustalili, że tempo deadenylacji wynosi 10 adenozyn/min. Ponadto zaproponowali uproszczoną metodę obliczania szybkości deadenylacji na podstawie zmiany wartości kwantyli dystrybucji. Dzięki tym narzędziom możliwe było uwidocznienie korelacji między tempem deadenylacji oraz sukcesywnej degradacji mRNA oraz pokazanie, że oba procesy zmieniają się w odpowiedzi na warunki stresowe. Co ciekawe, około 120 mRNA kodujących białkowe podjednostki rybosomu, które w swej masie reprezentują 40% transkryptomu, stanowią wyjątek i oprócz deadenylacji do swej degradacji wymagają także sprawnego eksportu z jądra komórkowego.
Pełna wersja artykułu pt. „Modeling of mRNA deadenylation rates reveal a complex relationship between mRNA deadenylation and decay”, opublikowanego w EMBO Journal, jest dostępna pod linkiem: https://www.embopress.org/doi/full/10.1038/s44318-024-00258-3.